近日,南京医科大学公共卫生学院马红霞教授、沈洪兵院士团队在国际期刊《美国人类遗传学杂志》(American Journal Of Human Genetics)发表题为Integrative splicing quantitative trait locus analysis reveals risk loci for non-small-cell lung cancer的研究论文。该研究通过剪接数量性状位点(splicing quantitative trait locus, sQTL)分析系统揭示了影响选择性剪接的遗传变异。在此基础上,整合非小细胞肺癌(non-small-cell lung cancer, NSCLC)全基因组关联研究(genome-wide association study, GWAS)数据,通过基于剪接的全转录组关联研究(splice-transcriptome-wide association study, spTWAS),识别NSCLC易感位点,探索遗传变异调控选择性剪接影响NSCLC易感性的生物学机制。(图1)
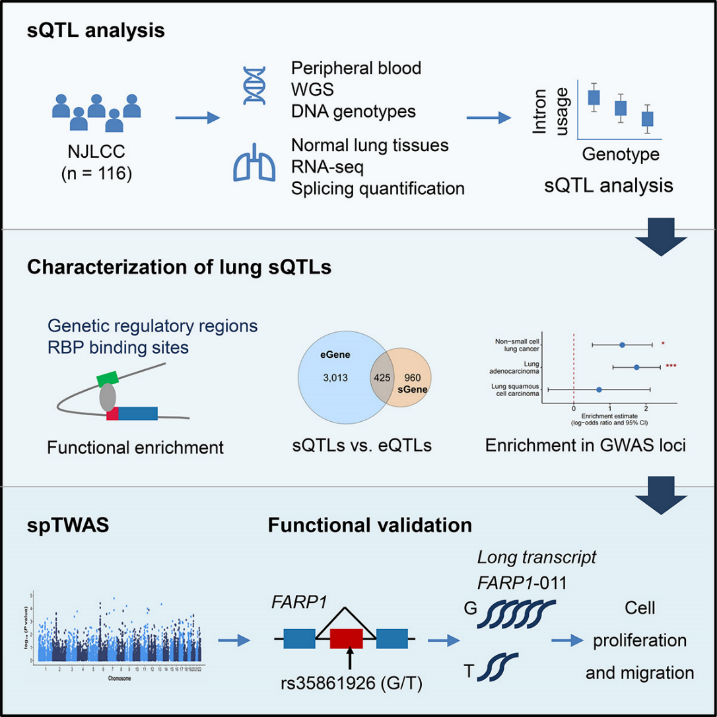
图1 研究设计流程图
肺癌是危害人类健康最严重的恶性肿瘤之一。约85%的肺癌为NSCLC。肺癌的发生是多因素多阶段的过程,受到环境和遗传因素的共同影响。此前,研究团队通过GWAS鉴定出数十个肺癌易感位点,在肺癌遗传易感机制研究方面取得了重大突破。然而,GWAS发现的肺癌易感位点仅能解释一小部分肺癌遗传度,并且大多数易感位点位于基因组非编码区,其生物学机制尚不清楚。选择性剪接通过选择性地使用剪接位点和外显子,使得单个基因可以产生多个成熟mRNA,是维持机体基因和蛋白质多样性的重要转录后调控机制,其通常受到遗传变异的调控。sQTLs和GWASs数据整合分析策略能够揭示出众多疾病易感位点的潜在生物学机制,sQTL在NSCLC发生过程中的作用目前尚不清楚。
本研究首先基于南京肺癌队列(Nanjing Lung Cancer Cohort,NJLCC)116名NSCLC患者的全基因组基因分型数据和正常肺组织转录组数据进行sQTL分析,鉴定出1,385个基因的选择性剪接事件受到sQTL调控。结果显示,60%的sSNPs(与选择性剪接关联性P值最低的遗传变异)位于剪接位点上下游10kb的范围内,此外57%的sSNPs位于相应选择性剪接事件所在基因;sSNPs显著富集于转录活性区和增强子区。随后,系统分析sQTLs与eQTLs之间的关系,发现大多数sQTLs效应独立于eQTLs,提示整合sQTLs和GWAS将有助于为NSCLC的病因学提供更多新见解。(图2)
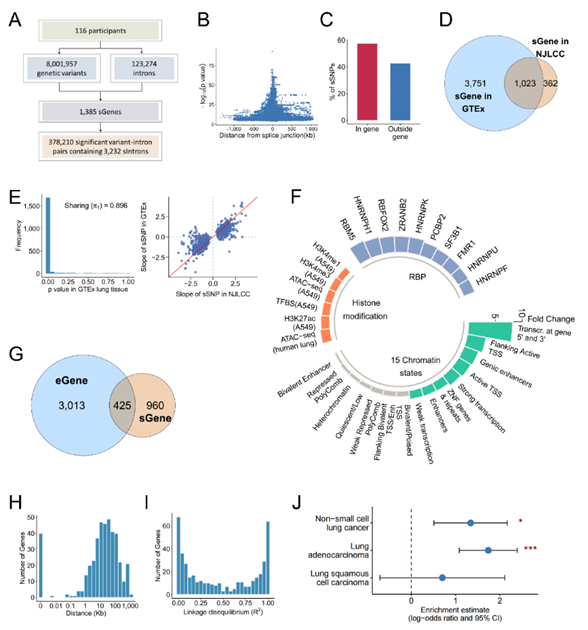
图2 sQTL的鉴定和基因组特征,以及sQTL和eQTL的比较
本研究进一步整合NJLCC基因型-选择性剪接数据库和非小细胞肺癌GWASs数据(包括13,327例NSCLC患者和13,328例对照),开展了基于剪接的全转录组关联研究(spTWAS),发现19个基因的23个选择性剪接事件可影响NSCLC发生风险。对共定位分析结果最为显著的EIF3E和FARP1基因进行了进一步探讨。染色体8q23.1区域EIF3E基因选择性剪接与肺腺癌发生风险显著相关,关联性P值最低的易感位点为rs443680(rs443680-G, OR = 1.09, 95%CI:1.05-1.14, P = 4.62×10-5)。rs443680与EIF3E基因选择性剪接事件的sSNP rs677031具有高度连锁不平衡关系(r2 = 0.99)。转录本数量性状位点和共表达分析发现,rs677031-G可下调内含子EIF3E:chr8:109,245,901-109,247,227的切除率,进而降低转录本EIF3E-011的表达水平(图3)。
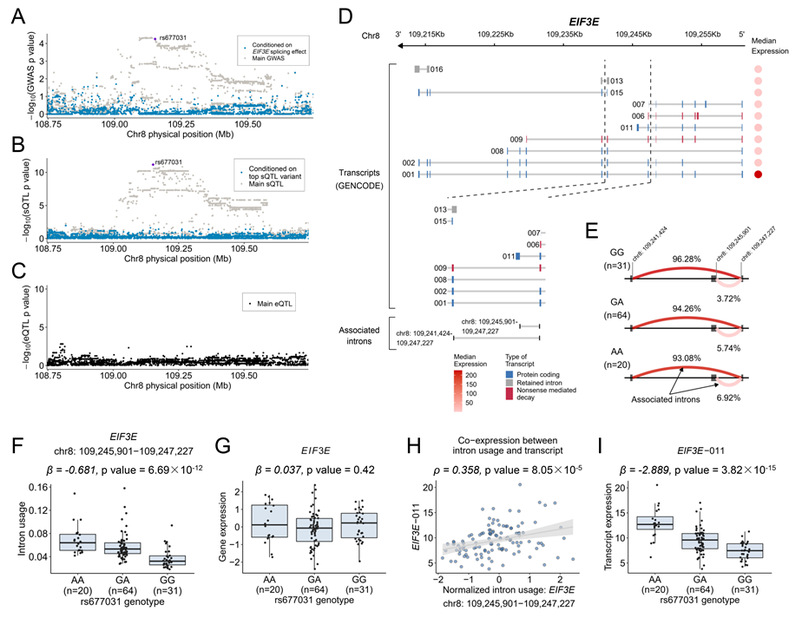
图3 易感位点调控EIF3E选择性剪接影响肺腺癌的发生风险
染色体13q32.2区域FARP1基因的选择性剪接事件(FARP1:chr13:99,090,112-99,091,058)也与肺腺癌发生相关(spTWAS Z = 4.28, P = 1.91×10-5),该区域GWAS关联性P值最低的易感位点为rs35861926(rs35861926-T, OR = 0.88, 95%CI:0.82-0.93, P = 1.87×10-5),且rs35861926也是FARP1基因选择性剪接事件的sSNP。进一步分析发现,rs35861926-T可下调内含子FARP1:chr13:99,090,112-99,091,058的切除率,进而降低转录本FARP1-011的表达水平。进一步的功能预测和分子生物学实验证实,rs35861926-T可促进FARP1第20号外显子跳跃,从而下调长转录本FARP1-011的表达水平,且该转录本可增强肺腺癌细胞系的增殖和迁移能力(图4)。
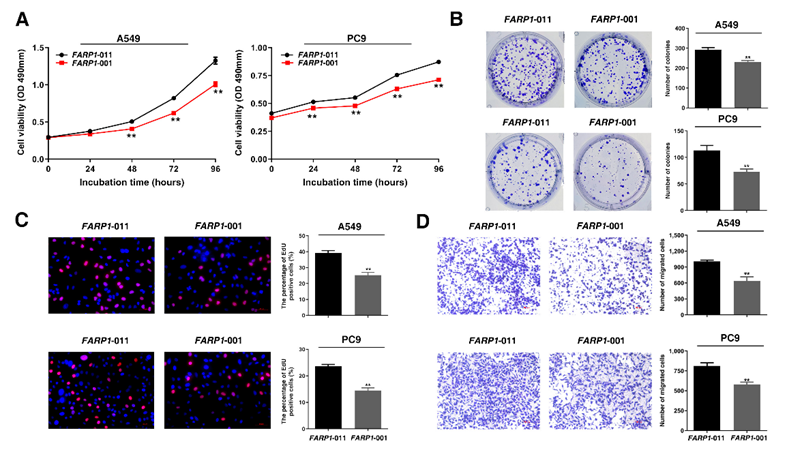
图4 长转录本FARP1-011促进肺腺癌细胞增殖和迁移
综上所述,本研究开展了一项全基因组sQTL研究,系统鉴定了中国人群肺组织中的sQTLs基因组特征,并通过spTWAS以及功能实验,为肺癌易感位点及其分子机制的解析提供了新的视角。
刊物:American Journal of Human Genetics
标题:Integrative splicing quantitative trait locus analysis reveals risk loci for non-small-cell lung cancer
原文链接:https://www.cell.com/ajhg/fulltext/S0002-9297(23)00248-3? journal=ajhg&publicationCode=ajhg&jc=ajhg
