近日,南京医科大学公共卫生学院陈峰教授课题组在《细胞报告》(Cell Reports)杂志在线发表了题为《非编码RNA与DNA甲基化在泛癌中的大规模整合》(Large-scale integration of the non-coding RNAs with DNA methylation in human cancers)的研究论著。公共卫生学院生物统计学系沈思鹏副教授为第一作者兼通讯作者,陈家进博士生为并列第一作者,赵杨教授为主要通讯作者,陈峰教授为资深作者。

癌症是一种复杂的疾病,表观遗传修饰中的DNA甲基化(DNA methylation)具有调节基因表达、基因组稳定性和影响细胞活性的功能,非编码RNAs(non-coding RNAs,包括miRNAs、lncRNAs、rRNAs、snRNAs和snoRNAs)可以在RNA水平上执行生物学调控。绘制非编码RNAs数量性状甲基化位点(ncRNA quantitative trait methylation site, ncQTM)效应,评估其在癌症和其他生物学过程中的因果作用具有重大的公共卫生意义与临床价值。
课题组将ncQTM分为两个模块进行研究:顺式(来自DNA甲基化位点的非编码RNA<=1Mb)和反式(来自DNA甲基化位点的非编码RNA>1Mb或染色体间的)。在癌症基因组图谱(TCGA)数据库中进行了32种癌症类型的8545名患者分析工作。并在临床蛋白质肿瘤分析联盟(CPTAC)中的763个肿瘤组织和基因型-组织表达(GTEx)中的516个正常组织进行了比较。
图1. 研究流程
在超过220亿关联分析中,最终确定了302,764个顺式ncQTM和79,841,728个反式ncQTM。其中在顺式结果中关联最多的非编码RNAs包括LINC00982, PSMB8-AS1, HOXA11-AS, HOXC-AS3和HOTAIR。在反式结果中关联最多的非编码RNAs包括PCED1B-AS1, LINC00355, RP11-356I2.4, DSCR4-IT1和ST8SIA6-AS1。此外,大多数反式关联(88.9%)为染色体间相关。
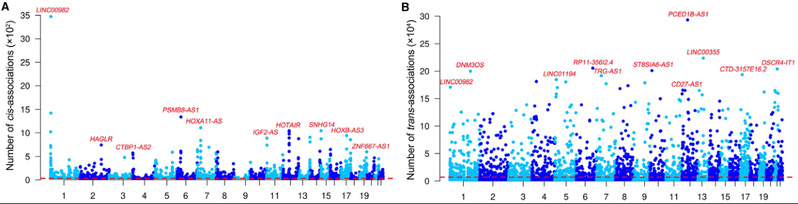
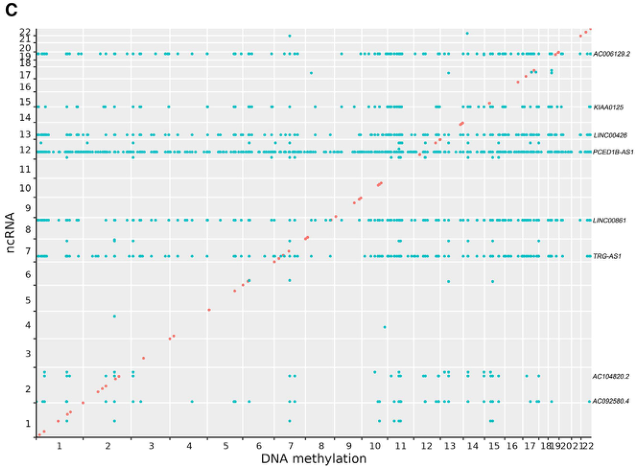
图2. 顺式与反式ncQTM的关联结果
为了高效利用32种人类癌症类型的ncQTM的综合资源,课题组开发了一个Pancan-ncQTM数据库:http://bigdata.njmu.edu.cn/Pancan-ncQTM/.

图3. Pancan-ncQTM数据库
时间:2023年3月
刊物:Cell Reports
标题:Large-scale integration of the non-coding RNAs with DNA methylation in human cancers
原文:https://doi.org/10.1016/j.celrep.2023.112261
